随着后基因组时代的到来和高通量测序技术的发展,产生了大量实验鉴定的蛋白质-蛋白质相互作用(protein-protein interactions, PPIs)数据,许多PPIs数据库也涌现出来,包括DIPs、BIND和HPRD等。PPIs在细胞结构和功能中发挥着重要的作用,网络结构的混乱会造成细胞生命活动的异常。深入研究PPIs对理解细胞生命活动、阐明疾病发病机制、药物设计和人类疾病防治等方面具有非常重要的意义。传统实验方法鉴定和识别PPIs耗费资源且周期较长,并且存在着噪声数据。因此,基于人工智能方法预测PPIs显得尤为重要,不仅可以预测未知的PPIs,还能对实验方法起到一定辅助作用,已经成为生物信息学的研究前沿和热点。
近日,数理学院人工智能与生物医学大数据研究团队于彬副教授,在计算机科学领域一区TOP期刊Expert Systems With Applications上发表标题为“Prediction of protein-protein interactions based on elastic net and deep forest”的研究论文。报道了基于深度森林预测蛋白质-蛋白质相互作用的人工智能算法模型—GcForest-PPI。该模型表现出较强的泛化能力以及优异的计算稳定性。于彬副教授为论文的第一作者及通讯作者,研究生陈成并列一作,青岛科技大学为第一完成单位。
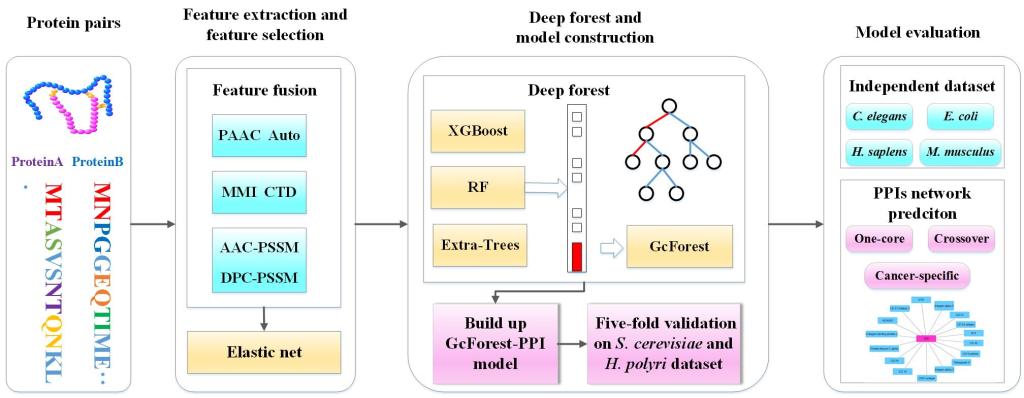
在蛋白质-蛋白质相互作用预测研究领域,本研究首次使用弹性网算法进行特征选择,剔除冗余特征和不相关的特征,降低了计算复杂度,提高运行效率。在该研究领域,我们首次使用深度森林预测PPIs,组合XGBoost、Random Forest和Extra-Trees构建深度森林模型(GcForest-PPI),通过深层结构挖掘序列特征和类别标签的非线性关系。S. cerevisiae和H. pylori数据集的结果表明基于树的深度学习方法具有较好的模型泛化能力。C. elegans、E. coli、H. sapiens以及M. musculus的数据集取得较好的预测效果,说明我们构建的模型能够进行跨物种预测,而且阐明了物种之间具有同源性。在CD9-core网络、crossover网络和cancer-specific网络数据集上验证了该方法预测PPIs网络的有效性。本文提出的GcForest-PPI人工智能算法模型可以成为生物信息学的一个有效工具,将为信号通路研究、致病基因挖掘、相互作用网络拓扑研究和人类疾病预防提供借鉴和指导。
文章链接:https://doi.org/10.1016/j.eswa.2021.114876


